非池化模型(No pooling)#
在complete pooled模型中,我们忽略了数据来自不同的被试这一事实
现在我们考虑另外一种情况,我们假设五个被试分别来自不同的分布,对五个被试进行不同的分析(每个被试的\(\mu_j\)都不一样,每个被试内的试次都服从均值为\(\mu_j\)的正态分布)
注意:我们假定,每个被试的数据之间完全没有关联,不同被试之间彼此独立。
从统计上讲,假定各被试之间的参数(例如均值μ)没有关系,或者说是完全异质。
# 设置绘图风格
sns.set(style="white")
# 创建子图
fig, axes = plt.subplots(1, 2, figsize=(14, 6))
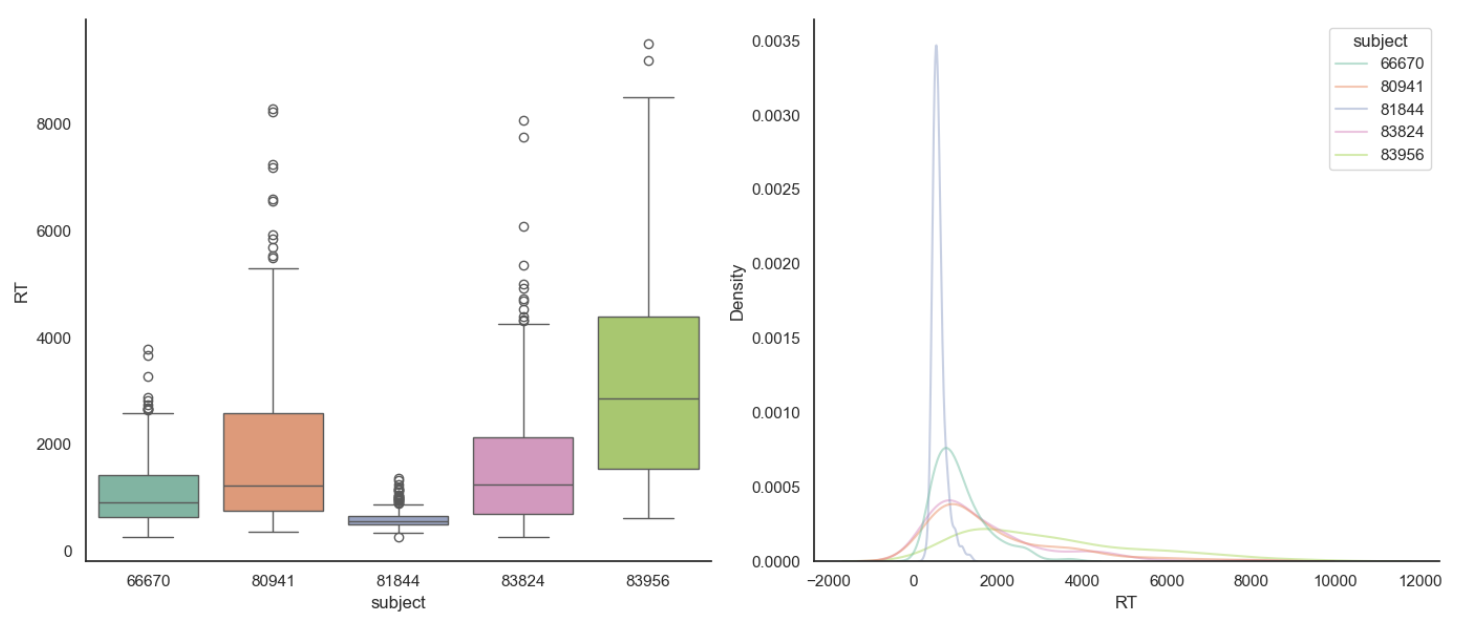
# 左侧箱线图:显示每个被试的反应时间分布
sns.boxplot(
data=df_first5,
x="subject",
y="RT",
palette="Set2",
ax=axes[0]
)
# 右侧核密度估计图:显示每个被试的反应时间密度分布
sns.kdeplot(
data=df_first5,
x="RT",
hue="subject",
palette="Set2",
common_norm=False,
alpha=0.5,
ax=axes[1]
)
# 显示图形
plt.tight_layout()
sns.despine()
plt.show()
Group-specific parameters
在完全池化模型中,我们使用了正态分布的参数来自总体层面
而在非池化模型中,我们认为正态分布的参数在组与组之间是不同的(group-specific) —- 可以认为每个被试的数据对应一个独立的正态分布模型
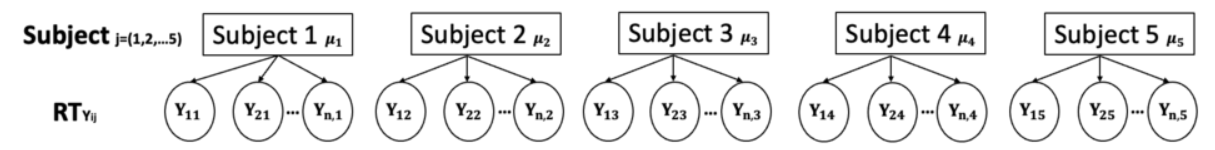
1.使用\(\mu_j\)来表示每个被试的反应时均值
不同被试的\(\mu_j\)不同
同一被试内的试次服从以\(\mu_j\)为均值的正态分布
2.同样,使用\(\sigma_j\)来表示每个被试内部每个试次的反应时变异性
和\(\mu_j\)类似,不同被试的\(\sigma_j\)不同
同一个被试服从以\(\sigma_j\)为标准差的正态分布
则对于被试 j 内的每个试次来说,反应时间满足:
模型定义及MCMC采样#
考虑到数据有5个被试,即j=1,2,3,4,5。因此,\(\mu_j和\sigma_j\)也有5个值。
在pymc中,我们可以通过定义坐标 coords 来实现
pm.Normal(..., dims="subject")此外,每个trial的数据来自于某位被试,因此可以通过
pm.MutableData("subject_id", mapped_subject_id, dims="obs_id")来定义每个trial数据 obs_id 和被试 subject 之间的映射。
# 建立被试 ID 映射表
subject_mapping = {subj_id: idx for idx, subj_id in enumerate(df_first5["subj_id"].unique())}
# 将被试 ID 转换为索引
mapped_subject_id = df_first5["subj_id"].map(subject_mapping).values
# 定义 pymc 模型坐标
coords = {
"subject": df_first5["subj_id"].unique(),
"obs_id": df_first5.index.values
}
with pm.Model(coords=coords) as no_pooled_model:
# 对 RT 进行 log 变换
log_RTs = pm.MutableData("log_RTs", np.log(df_first5['RT']))
# 定义被试特定的均值和标准差
mu = pm.Normal("mu", mu=7.5, sigma=5, dims="subject")
sigma = pm.Exponential("sigma", 1, dims="subject")
# 定义观测数据的映射 (obs_id -> subject)
subject_id = pm.MutableData("subject_id", mapped_subject_id, dims="obs_id")
# 定义观测值 (obs_id 映射到对应 subject 的 mu 和 sigma)
y = pm.Normal("y_est", mu=mu[subject_id], sigma=sigma[subject_id],
observed=log_RTs, dims="obs_id")
# MCMC 采样
no_pooled_trace = pm.sample(1000, return_inferencedata=True)
pm.model_to_graphviz(no_pooled_model)
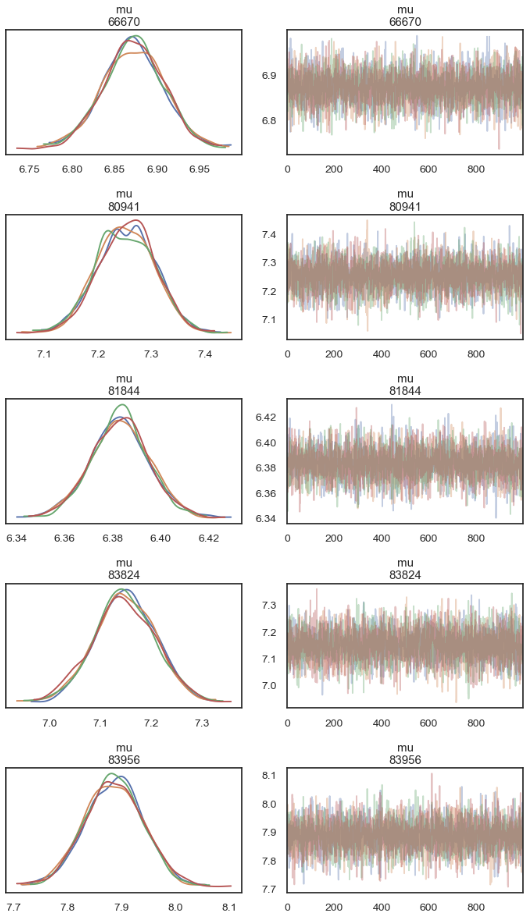
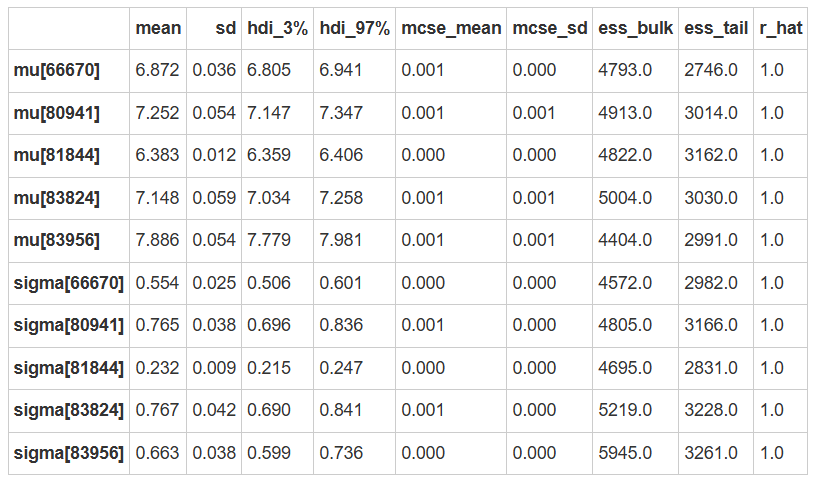
查看后验参数估计#
可以发现,对于每个被试,均有不同的参数值(包括\(\mu和\sigma\))
ax = az.plot_trace(
no_pooled_trace,
var_names=["mu"],
filter_vars="like",
compact=False,
figsize=(7,12))
plt.tight_layout()
az.summary(no_pooled_trace)
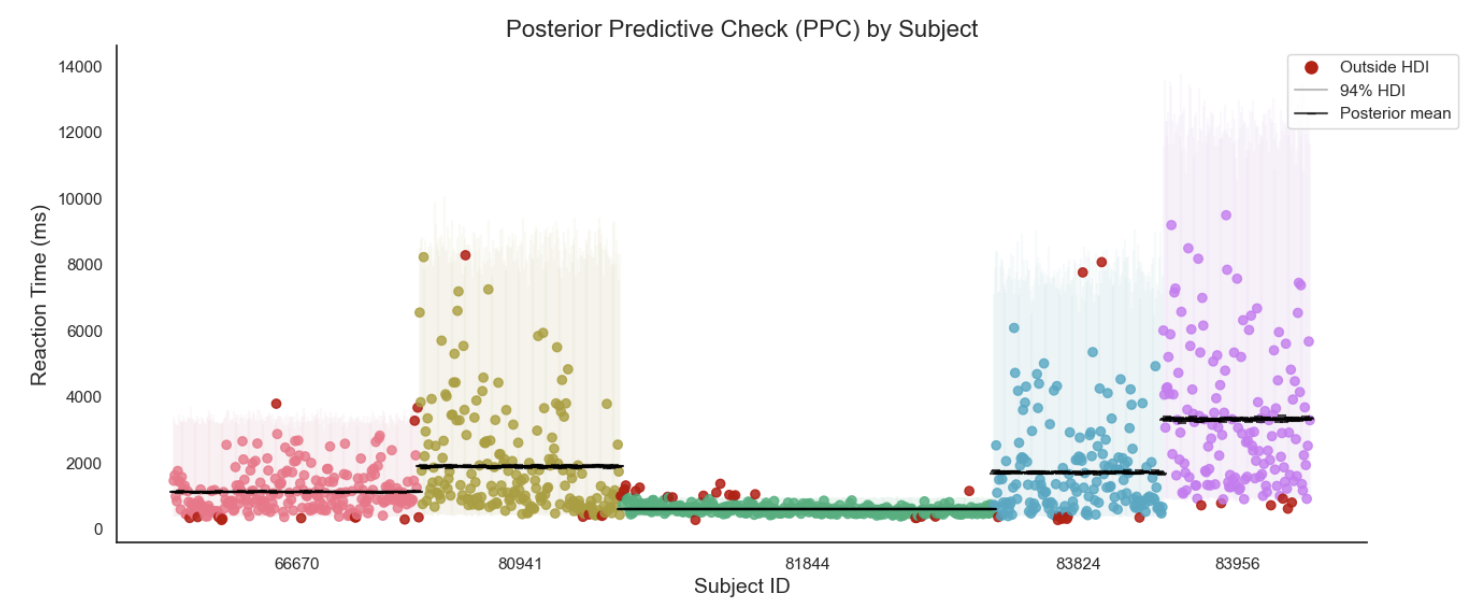
后验预测分布#
no_ppc = pm.sample_posterior_predictive(no_pooled_trace,
model=no_pooled_model)
no_hdi_sum = ppc_sum(ppc = no_ppc,
data=df_first5)
进行对数转换:
no_hdi_sum = inv_log_hdi_sum(no_hdi_sum)
ppc_plot(hdi_sum=no_hdi_sum)
非池化模型的缺点#
可以看出,每个组的均值和方差都是不同的,它针对每一个被试都建立了一个独特的模型,能够很好地捕捉个体差异。
但是,这样做存在一些缺陷:
在小样本数据上,非池化模型存在过拟合的风险。它不仅拟合了数据特点,还拟合了每个被试独特个性的特点。
非池化模型假设每个组都属于不同的分布,因此其得出的结果难以用来预测新组别的情况。例如,新加入一个被试,我们很难选择某个模型对其进行预测。